La única imagen que aparece en “El origen de las especies” de Charles Darwin (1809-1882) es un árbol filogenético (Fig. 1).
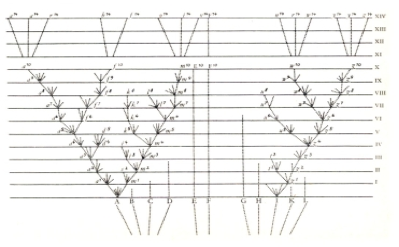
Bien sabía él la importancia de este árbol que representa la historia evolutiva de los seres vivos. Y es que Charles Darwin equiparó a cada especie con una fuente de información tal que le permitía construir genealogías basadas en las características fenotípicas o la morfología.
Un siglo después, el biofísico y microbiólogo Carl Richard Woese (1928-2012) decidió emplear otra herramienta, muy poderosa, para tal reconstrucción genealógica. Ésta le permitiría obtener más información referente a las relaciones evolutivas entre seres vivos, incluyendo los microscópicos de sosa morfología.
Lo que caracteriza a los sistemas biológicos es la biodiversidad y el DNA su sustrato.
La genialidad de Carl Woese fue ver a la célula como documental de información formidable, lo suficientemente útil como para reconstruir el árbol genealógico y la película misma de la vida. Esto se puede hacer, según palabras de Woese, mediante el uso del “registro fósil interno de la célula”, siempre que se acuda al marco conceptual básico de la biología: la teoría evolutiva. De acuerdo a nuestras nociones darwinianas, la biodiversidad es el resultado de un proceso evolutivo. Lo que caracteriza a los sistemas biológicos es la biodiversidad y el DNA su sustrato, concluyó Carl Woese. Según Norman Pace, “nadie más que Woese estaba pensando en esta dirección en aquel entonces, como para buscar evidencias de la diversidad de la vida basadas en la secuencia del DNA” (Arnold 2014). Carl fue más allá: como todos los organismos, por muy simples que sean, poseen ribosomas y la traducción del RNA ribosomal (rRNA) a proteínas en ribosomas es una característica universal, ancestral y esencial (Sapp 2013), entonces debía emplear la secuencia de rRNA como herramienta para construir el árbol de la vida. En efecto, en la vereda de la evolución biológica, las novedades evolutivas están registradas en el material genético.
Vale la pena seguir al menos tres de las premisas planteadas por Woese que le llevaron a construir la genealogía de los seres vivos aprovechando una biomolécula: el rRNA. La primera y más obvia es que no es posible comparar en términos morfológicos organismos tan disímiles como bacterias y delfines, no sólo porque median 6 órdenes de magnitud entre el tamaño de uno y de otro, sino porque ¡no hay estructuras que comparar!. Segunda, si bien a los seres vivos nos define y caracteriza, digamos, un tipo de metabolismo básico para transformar la materia y obtener y usar la energía, al compararnos sólo podemos formar “grupos metabólicos”, dentro de los cuales las bacterias, a diferencia de los delfines, estarán presentes en todos, por ser el dominio celular de mayor diversidad metabólica, como también lo demostrara Woese. Además, los “grupos metabólicos” informan sobre procesos biogeoquímicos que desatan relaciones ecológicas, más que sobre relaciones evolutivas. Y, en definitiva, como el metabolismo es la expresión de la información genética, Woese decidió que lo más atinado era comparar la información genética de los seres vivos “¿Toda la información genética?” No, claro que no: parte del genoma sí, y esta “parte” definitivamente no debía involucrar a los genes implicados en la síntesis de enzima que participan en rutas metabólica específicas en los tan mencionados “grupos metabólicos” (por ejemplo, en la respiración anaerobia vs. la aerobia).
Conocer la historia de los ribosomas y el rRNA, para aprender algo de la historia de los seres vivos.
Lo que sí es común a todos los seres vivos es la traducción del rRNA a proteínas. Carl Woese lo supo y lo exploró: “conocer la historia de los ribosomas y el rRNA, para aprender algo de la historia de los seres vivos”. La selección de rRNA se sumó a sus inquietudes en torno a la evolución de los sistemas de traducción a proteínas, básicamente del aparato traductor y el código genético. Más aún, para Carl Woese esta biomolécula que guarda información, el RNA, es un cronómetro universal, un reloj evolutivo con latido regular, el tic-tac-tic-tac de los cambios o mutaciones aleatorias. Ésta no es una aportación cualquiera, es una manera mediante la cual se puede “ajustar” o “compaginar” la tasa de cambios moleculares y los eventos geológicos, tal y como Emile Zuckerkandl y Linus Pauling (1962) lo habían descrito usando una proteína (la hemoglobina). Y para colmar las aportaciones de un Woese clavado, clavadísimo, en el estudio del RNA, podemos afirmar que él es padre de la teoría del “Mundo del RNA” (1967), cuya hipótesis central es que, dadas las propiedades autorreplicantes del RNA, que resguarda información genética (como el DNA) y actúa como catalizador (proteína enzimática), éste ácido nucleico es el precursor de los sistemas vivos. Cabe decir que se trata de una hipótesis muy fructífera y manantial de investigaciones diversas que derivaron en hipótesis más completas sobre el origen de la vida.
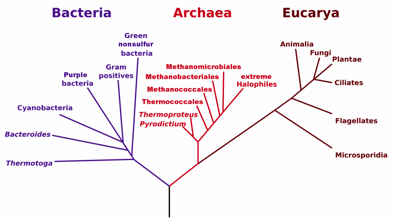
Después de hacer un análisis comparativo de secuencias moleculares accesibles que cubrieran lo más completamente posible la vasta variedad de sistemas vivos, Woese y su estudiante postdoctoral George E. Fox, encontraron las relaciones evolutivas de los seres vivos (1977). Ahora ya sabemos que estamos representados en tres dominios celulares: Bacteria, Archaea y Eukarya (Fig. 2). Hubo un tiempo en el cual todo biólogo soñaba con descubrir una nueva especie; Woese no soñó (creo yo) ni imaginó descubrir un nuevo dominio celular, el Archaea… y una que otra Archaea de epíteto “woesiano”: Pyrococcus woesei y Methanobrevibacterium woesei.
Resulta de lo más complicado refutar los resultados obtenidos por Woese, particularmente porque empleó una biomolécula diversificada en todos los seres vivos. Su meditada y relevante selección lo llevó a optar por uno de los genes más antiguos, el rRNA, pero también el más estable, que se ha copiado generación tras generación de bacterias, hongos o tortugas, con un elevado grado de precisión y que ha mutado muy lentamente; mutaciones que hasta parecieran responder a eventos de especiación, es decir, mutaciones que parecieran coincidir con el no explicado por Darwin, origen de las especies.
Pero la historia no fue del todo alegre en un primer momento… un largo periodo de tiempo, de hecho.
Los resultados sobre filogenias moleculares de Carl Woese no fueron aceptados sino casi 20 años después, ya que en aquellas alegres décadas (1970-1980) “la biología molecular parecía más una disciplina de ingeniería, ajena y alejada de la teoría de la evolución” (Sapp 2014). Los ingenieros genetistas conocían una técnica de extremo cuidado, no así el fundamento de las teorías biológicas; ante los resultados de Woese era de esperarse que despreciaran el hallazgo de un microbiólogo que osó utilizar las técnicas y los datos de su disciplina de élite. Los disparates de la ingeniería genética sin un “background” evolutivo y ecológico conlleva a atrocidades tales como la Revolución Verde (y el concomitante uso de enormes cantidades de agua y, peor aún, agroquímicos).
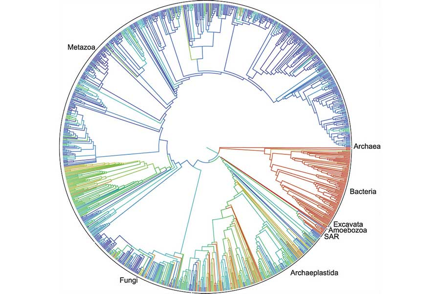
Conforme fue conociéndose el trabajo de Woese, se realizaron nuevos análisis; lo reproducible es sólo creíble (¿Cómo el origen de la vida?), y “con los datos de secuencias génicas accesibles a más y más investigadores, se hizo evidente para casi todo aquel relacionado con biología que el árbol de la vida de Carl Woese era, de hecho, correcto” (Arnold 2014). Siempre he creído que los bioinformáticos nos salvarían al ayudar a sistematizar la enorme cantidad de información que la biología e ingeniería molecular están generando a cada instante. Y sí, lo están logrando haciendo gala de un regio esfuerzo en colaboración, abierto y accesible. El pasado 19 de septiembre de 2015, salió a la luz pública el árbol de la vida, construido mediante filogenias, publicadas junto con clasificaciones taxonómicas de ca. 2.3 millones de especies biológicas conocidas, pertenecientes a los tres linajes descritos por Woese (Fig. 3). Éste forma parte del proyecto Open Tree of Life, un sistema abierto que facilita la contribución de la comunidad científica internacional, y permitirá que el árbol se actualice continuamente… Y vaya que debe actualizarse, pues el árbol resultante dista enormemente de representar la diversidad “real”, al estar sobrerrepresentados los grupos de animales metazoarios y de plantas y algas (Archaeplastida o Primoplantae), por sobre la microbiota, pobremente analizada. Malo, pues tal y como Woese también lo demostró, los microorganismos representan la vasta mayoría de diversidad genética, metabólica y de nichos ecológicos, y se incluyen en los tres dominios celulares (p.e., los amoebozoa o amibas de la Fig. 3; microorganismos eucariontes). Lo destacado y aplaudible, sin duda alguna, es que se ha abierto una plataforma que permitirá que, en esencia, nuestra compresión sobre el surgimiento y devenir de la vida, y el árbol de Woese crezcan, llenándose de ramas, ramotas, y ramitas.
Así, los árboles filogenéticos nos permiten cuantificar las relaciones evolutivas entre seres vivos y adentrarse en las profundidades de los tiempos para comprender cómo ha iniciado y evolucionado la vida, y cómo la biodiversidad en nuestro planeta está estructurada en términos ecológicos y genéticos, y con capacidades y potencialidades incluso biotecnológicas. Woese apreció en cada célula no una foto sino una película, un sistema con información aparentemente críptica que narra su propia novela y la de sus ancestros. Dentro de éstas, una ancestral molécula “informacional”, universal y estable fue aprovechada cual “código de barras”, para comparar a todo los seres vivos y re-escribir una historia con más de 3.7 mil millones de años en su haber.
Carl Woese eligió la molécula correcta, y eso no fue casual. C2











maria porras -
Buenas noches, muchas gracias por el ensayo! pero tengo una duda, por qué Woese estaba en contra de la endosimbiosis?porque el consideraba que LUCA ocurrió sin necesidad de ese proceso o por qué?Graciaas.